超高速原子力显微镜HS-AFM+质量光度计观察粘连蛋白介导DNA环挤出
超高速原子力显微镜HS-AFM+质量光度计观察粘连蛋白介导DNA环挤出
超高速视频级原子力显微镜(High-Speed Atomic Force Microscope,HS-AFM)由日本 Kanazawa 大学 Prof. Ando 教授团队研发,日本RIBM公司(生体分子计测研究所株式会社,Research Institute of Biomolecule Metrology Co., Ltd)商业化的产品,可以达到视频级成像的商业化原子力显微镜。HS-AFM突破了传统原子力显微镜“扫描成像速慢"的限制,能够在液体环境下超快速动态成像,分辨率为纳米水平。样品无需特殊固定,不影响生物分子的活性,尤其适用于生物大分子互作动态观测。超高速视频级原子力显微镜HS-AFM主要有两种型号,SS-NEX样品扫描(Sample-Scanning HS-AFM)以及PS-NEX探针扫描(Probe-Scanning HS-AFM)。推出至今,已有150多位用户,发表 SCI 文章 300 余篇,包括Science, Nature, Cell 等杂志。
相较于目前市场上的原子力显微镜成像设备,HS-AFM突破了 “扫描成像速慢"的限制,扫描速度高可达 20 frame/s,并且有 4 种扫描台可供选择。样品无需特殊固定染色,不影响生物分子的活性,尤其适用于生物大分子互作动态观测。液体环境下直接检测,超快速动态成像,分辨率为纳米水平。探针小,适用于生物样品;悬臂探针共振频率高,弹簧系数小,避免了对生物样品等的损伤。悬臂探针可自动漂移校准,适用于长时间观测。采用动态PID控制,高速扫描时仍可获得清晰的图像。XY轴分辨率2nm;Z轴分辨率0.5nm。
HS-AFM不仅拥有超高扫描速率与原子级别分辨率,而且具有操作的简易性,使得对单分子动态过程的捕捉变得十分方便,为科研工作者研究和理解生物物理、生物化学、分子生物学、病毒学以及生物医学等领域的单分子动态过程提供了一款强大的工具。全新的HS-AFM采用了新的高频微悬臂架构,更低噪音、更高稳定性的控制器,高速扫描器,缓冲防震设计,主动阻尼,动态PID,驱动算法优化,多种前沿技术,可以实现在超高速下获取高分辨的生物样品信息。新系统整合了基于工作流程的操作软件,直观的用户界面与流程化、自动化的设置使得研究人员可以专注于实验设计,不需要复杂的操作和条件设置,快速获取数据,加速研究的产出。

基因组DNA折叠形成环状以及拓扑关联结构域(Topologically associating domains,TADs),从而组成了基因组复杂的三维结构,这些三维结构具有重要的结构和调控作用。先前的研究已经逐渐揭示出基因组结构是由染色体结构维持SMC(Structural maintenance of chromosomes)蛋白复合物所介导的环挤出(DNA loop extrusion)过程形成的(图1)。单分子层面的研究表明SMC蛋白复合体和Cohesin蛋白的确能够将DNA挤压形成环状。因此,关于基因组的三维结构形成研究者们提出了“环挤出假说",认为染色体结构维持的SMC复合物组织就基因组的拓扑结构,并通过环挤出来实现这一功能。但是这一过程的具体细节是如何进行的还不得而知。
奥地利维也纳生物中心 (VBC)分子病理学研究所Jan-Michael Peters研究组发现粘连蛋白通过“摆动和钳夹"机制介导 DNA 环挤出。2021年10月7日出版的《细胞》杂志发表了这一成果。作者分析了人类粘连蛋白-NIPBL 复合物如何介导环挤出,并使间期细胞中的染色质折叠。 他们已经确定了环挤出所需的 DNA 结合位点和大规模构象变化,并确定了这些是如何协调的。他们的结果表明 DNA 通过自发的 50 nm 摆动的粘连蛋白铰链转移,将 DNA 交给 SMC3 的 ATPase 头部,在那里结合 ATP,然后DNA 被 NIPBL 夹住。
在这个过程中,NIPBL 从铰链“跳跃"到 SMC3 头部,从而可能将自发铰链摆动与 ATP 依赖性 DNA 夹紧结合起来。 这些结果揭示了粘连蛋白-NIPBL 和可能的其他染色体结构维持 (SMC)复合物如何介导环挤出的机械原理。
该实验过程通过借助日本RIBM公司研发的超高速视频原子力显微镜HS-AFM来完成,HS-AFM突破了传统原子力显微镜“扫描成像速慢"的限制,能够实现在液体环境下超快速动态成像,分辨率为纳米水平。待测样品无需特殊固定,不影响生物分子的活性,尤其适用于生物大分子互作动态观测。
目前,一些体外的生化重构实验一定程度上已经证实了环挤出假说,这些结果证明SMC蛋白与Cohesin蛋白会以2.1kb/s的速度挤压DNA,并且这一过程依赖于ATP酶活性(图2)。但限于实验分辨率等问题,环挤出的过程是如何实现的具体细节还不得而知。
DNA环挤出过程中具体的构象变化是如何发生的呢?为了揭开这一过程的全貌,作者们应用了高速原子力显微镜(High-speed atomic force microscopy,HS-AFM)对Cohesin进行了实时成像。
在ATP存在的情况下的,Cohesin三聚体复合物中会在环状、杆状以及弯曲状构象之间的变化。作者们发现DNA是通过Cohesin蛋白铰链的弯曲进而易位的,这样将DNA转移到SMC3的ATP酶活性头部,在该位置与ATP结合,DNA被NIPBL夹住。在此过程中NIPBL从铰链向SMC3头部移动,引发铰链区域的自发摆动以及ATP依赖的DNA结合,从而形成一个循环,使得DNA从铰链延伸到SMC3的头部,在此循环周期的末尾,DNA片段可以转移到SMC1的头部。由此,作者们提出了DNA环挤出的“摆-夹"“Swing and clamp"模型,这类似于抓娃娃机的小爪子,可以将通过铰链的弯曲伸出机械手“抓住"DNA,然后上提,从而通过一次次循环促使DNA逐步环挤出。
结果:
(1):人黏连蛋白由四个亚基组成:SMC1,SMC3,Scc1和STAG1。SMC1和SMC3通过它们的铰链结构域异二聚化。SMC1-SMC3铰链异二聚体与STAG1的“ U"的底部直接接触,DNA首先由NIPBL和STAG1募集到处于SMC1和SMC3头部结构域分离状态的黏连蛋白,ATP结合后,Scc1连接SMC1和SMC3的ATPase头部结构域形成环,并将DNA捕获在SMC隔室内。然后,NIPBL和STAG1包裹在DNA周围,形成中ZHONG央通道并进一步包裹DNA。
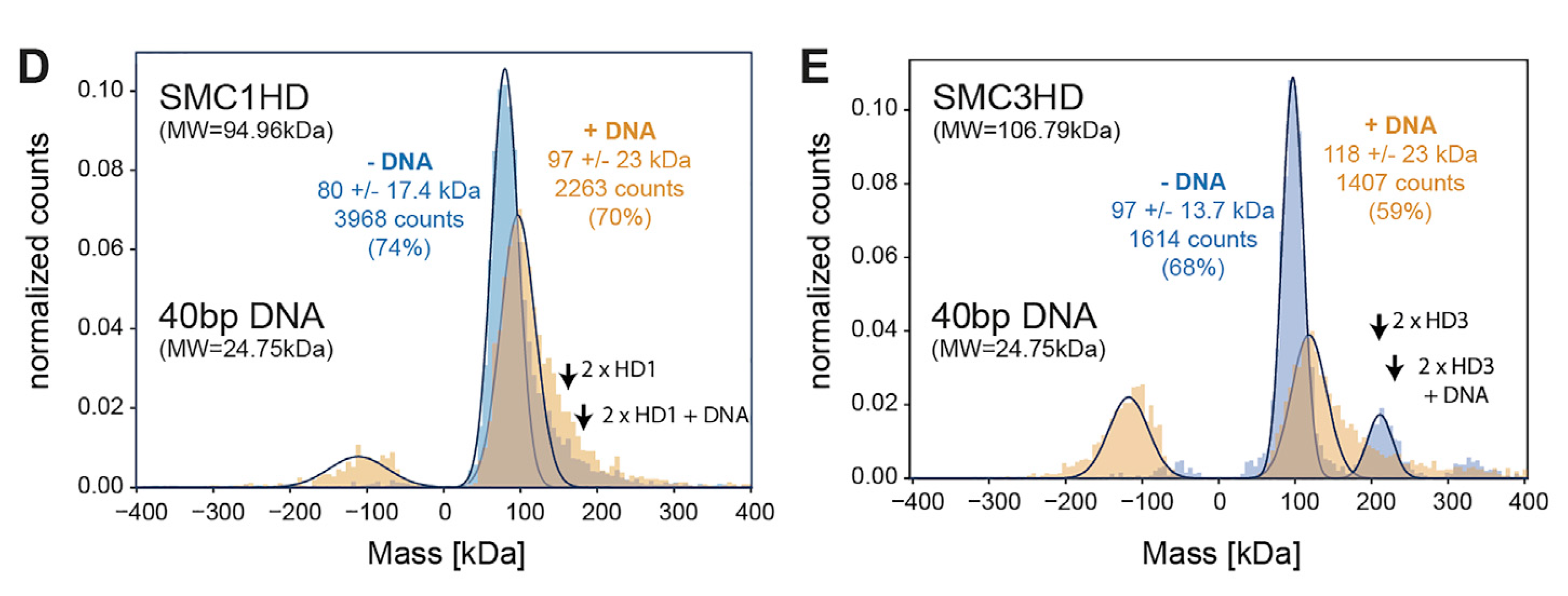
(2):通过TwoMP质量光度法(单分子质量光度计分析系统)证实了SMC1和SMC3头部结构域(SMC1HD、SMC3HD)可以各自结合单个DNA分子作为单体:在存在ATP的前提下,对SMC1HD和SMC3HD含和不含40碱基对(bp)双链(ds)DNA进行质量光度分析,负质量对应于蛋白质-DNA复合物在两帧之间从表面解离的事件。
(3):冷冻电镜Cryo-EM在不同的构象中观察到了黏连蛋白,但尚不清楚哪些在天然条件下存在,以及黏连蛋白如何从一种构象变化到另一种构象。因此,作者通过超高速视频级原子力显微镜HS-AFM实时成像黏连蛋白。为了确定哪些亚基和结构域可以通过HS-AFM可视化,作者分析了在含有SMC1、SMC3和SCC1的三聚体及结合到NIPBL(N)的三聚体,以及含有SMC1、SMC3和SCC1和STAG1(S)的四聚体及结合到NIPBL的四聚体,图F中显示了对应于铰链(hi)、头部(he)、NIPBL(N)和STAG1(S)的密度。

(4):随后通过质量光度法测定的二聚体(SMC1、SMC3)和三聚体(SMC1、SMC3和SCC1)的质量分布,可以证实这些复合体中含有SCC1,并且二聚体(286 kDa)和三聚体(399 kDa)的理论质量与测量值非常一致(图G、H)。质量光度法(Mass Photometry)是一种革命性的分子分析新方法。它能够准确测量溶液中天然状态下的单个分子的质量数,无需标记,为生物分析和生物分子功能研究开辟了新的途径。其原理是:粒子散射的光信号与粒子的体积和折射率成线性关系。由于生物分子的光学性质和密度仅有百分之几的差别,所以散射信号也就与分子的质量成正比,因此可以用光称量单个分子。散射信号与质量的相关性适用于各种生物分子(例如糖蛋白、核酸和/或脂质),这使得质量光度法成为溶液中生物分子的通用分析工具。

(5):在ATP存在下,(SMC1、SMC3和SCC1)三聚体复合物方式上在环状(rings)、棒状(rods)和扭曲(bent)构象之间交替出现(图I)。在环形复合体中,卷曲的线圈被拉伸但彼此分开,而头部结构域要么相互接触,代表ATP接合状态;要么头部结构域脱离,代表ATP非接合状态。在杆状复合体中,头部结构域脱离,线圈对齐;在许多情况下,相互扭曲(图I),在这种状态下,铰链有时会短暂打开。

(6):(SMC1、SMC3和SCC1)三聚体复合物在接合和脱离状态之间振荡,在后一种构造中,可以对齐和弯曲其线圈(图K,head movement,coil alignment);当STAG1和NIPBL与三聚体结合时,也观察到类似的变化;当线圈折叠成弯曲是不对称状态时,铰链靠近一个头部,但不靠近另一个头部(图K,hinge bending,铰链弯曲)。
讨论:
由染色体结构维持SMC蛋白复合物所介导的DNA环的挤出过程仍然是一个迷,这一过程被认为具有重要的结构和调控功能。作者通过超高速视频级原子力显微镜HS-AFM实时成像技术首SHOU次深入了解了人类粘连蛋白-NIPBL复合物易位DNA以介导DNA环挤出的机制原理,并可能对理解其他SMC复合物如何发挥这一功能具有更广泛的意义。
作者通过超高速视频级原子力显微镜实时成像,实现了对Cohesin促使的DNA环挤出过程的全纪录,从而能够将DNA从一个结合位点带到另一个结合位点,揭开了SMC复合体进行基因组的折叠的以及促进复杂基因组三维拓扑结构形成的机制。通过质量光度法测定的二聚体(SMC1、SMC3)和三聚体(SMC1、SMC3和SCC1)的质量分布。
参考文献:
Benedikt W. Bauer, et al. Cohesin mediates DNA loop extrusion by a“swing and clamp"mechanism. Cell 184, 5448–5464, October 14, 2021.
新品推荐——日本RIBM公司研发的超高速视频原子力显微镜HS-AFM来到中国
为了更好地服务国内客户,北京佰司特科技有限责任公司将这款超高速视频级原子力显微镜引进中国,如果您有科研上的需要,欢迎致电联系我们!
北京佰司特科技有限责任公司
类器官串联芯片培养系统—HUMIMIC;类器官灌流式培养和代谢监测系统—IMOLA;
蛋白稳定性分析仪—PSA-16;单分子稳定性分析仪(磁镊力谱测量仪)—HiMT;单分子质量光度计—TwoMP;超高速视频级原子力显微镜—HS-AFM;微流控扩散测量仪—Fluidity One-M;
微纳加工点印仪—NLP2000/DPN5000;台式原子力显微镜—ACST-AFM;全自动半导体式细胞计数仪—SOL COUNT;农药残留定量检测仪—BST-100;

